ex7:k-means and PCA
前半
概述
在这个练习中,你将实现K均值算法并将其用于图像压缩。首先,你将在一个2D示例数据集上开始,这将帮助你直观地了解K均值算法的工作原理。之后,你将使用K均值算法进行图像压缩,通过将图像中出现的颜色数量减少到仅包含那些在该图像中最常见的颜色。在这一部分的练习中,你将使用ex7.m。
必要的文件如下:
ex7.py - 用于K均值算法第一练习的Python脚本ex7data2.mat - K均值算法的示例数据集bird_small.png- 示例图像 displayData.py - 显示存储在矩阵中的2D数据的函数plotDataPoints.py- K均值聚类中质心的初始化函数plotProgresskMeans.py- 绘制K均值每一步的函数runkMeans.py - 运行K均值算法
需要完成的文件:
findClosestCentroids.py- 寻找最接近的质心(在K均值中使用)computeCentroids.py- 计算质心均值(在K均值中使用)kMeansInitCentroids.py - K均值聚类的质心初始化
导入
1
2
3
4
5
6
7
8
9
10
| import matplotlib.pyplot as plt
import numpy as np
import scipy.io as scio
from skimage import io
from skimage.util import img_as_float
import runkMeans as km
import findClosestCentroids as fc
import computeCentroids as cc
import kMeansInitCentroids as kmic
|
Scikit-image(skimage)是一个基于SciPy库的图像处理库,提供了一系列用于图像处理的工具和算法。它包含了许多常用的图像处理任务的实现,如图像过滤、边缘检测、形态学操作、图像变换等。
寻找质心
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
|
print('寻找最近的质心。')
data = scio.loadmat('ex7data2.mat')
X = data['X']
k = 3
initial_centroids = np.array([[3, 3], [6, 2], [8, 5]])
idx = fc.find_closest_centroids(X, initial_centroids)
print('前3个示例的最近质心:')
print('{}'.format(idx[0:3]))
print('(应分别为0, 2, 1的最近质心)')
input('程序暂停。按回车键继续')
|
这里需要完成findClosestCentroids.py
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
| import numpy as np
def find_closest_centroids(X, centroids):
K = centroids.shape[0]
m = X.shape[0]
idx = np.zeros(m)
for i in range(m):
distances_squared = np.sum((X[i] - centroids) ** 2, axis=1)
closest_centroid_index = np.argmin(distances_squared)
idx[i] = closest_centroid_index
return idx
|
虽然centroids是一个$(3 \times 2)$的数组,而X[i]是一个一维的数组,但是两者之间依然可以加减,X[i]会复制成3列,然后减去centroids,这是numpy的特性:广播运算
指定axis=1,在numpy,是指定行的运算,结果即是数据点到质心之间距离的平方,distances_squared是一个一维数组
最后使用 np.argmin函数,找出数组中最小值的索引,放入到idx中
移动质心
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
print('计算质心均值。')
centroids = cc.compute_centroids(X, idx, k)
print('在找到最近质心后计算的质心: \n{}'.format(centroids))
print('应为')
print('[[ 2.428301 3.157924 ]')
print(' [ 5.813503 2.633656 ]')
print(' [ 7.119387 3.616684 ]]')
input('程序暂停。按回车键继续')
|
完成compute_centroids 函数
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
| import numpy as np
def compute_centroids(X, idx, K):
(m, n) = X.shape
centroids = np.zeros((K, n))
for i in range(K):
points_in_cluster = (idx == i)
if np.any(points_in_cluster):
centroids[i] = np.mean(X[points_in_cluster], axis=0)
return centroids
|
这里需要遍历每个质心,计算属于它的所有点的平均值。
在for循环中,points_in_cluster = (idx == i)会得到一个大小和idx一样的布尔数组
centroids[i] = np.mean(X[points_in_cluster], axis=0),则将布尔数组作为索引,成功地找到了每个质心对应的数据点,而将其他的数据点置为0,这一个方法在逻辑回归和SVM中已经用过多次
最后指定axis=0,即指定按列运算,算出x,y坐标的均值,得到质心移动的位置
K均值聚类
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
|
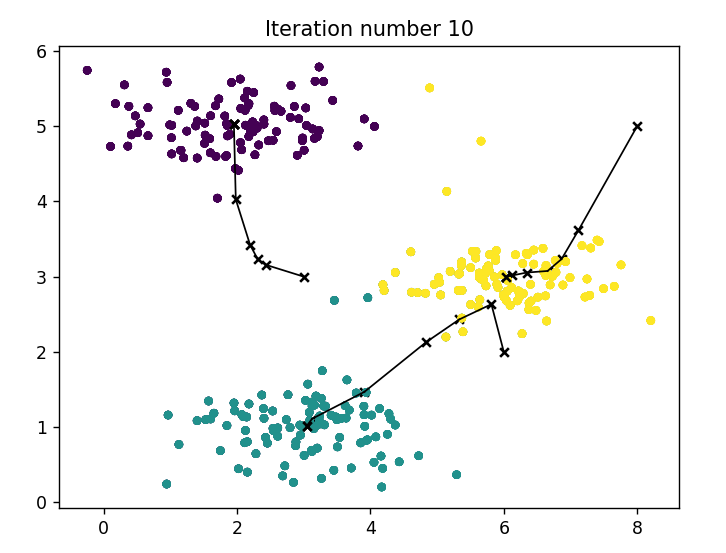
print('在示例数据集上运行K均值聚类。')
data = scio.loadmat('ex7data2.mat')
X = data['X']
K = 3
max_iters = 10
initial_centroids = np.array([[3, 3], [6, 2], [8, 5]])
centroids, idx = km.run_kmeans(X, initial_centroids, max_iters, True)
plt.show()
print('K均值完成。')
input('程序暂停。按回车键继续')
|
使用K均值聚类,画出质心移动的图像
图像的聚类
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
|
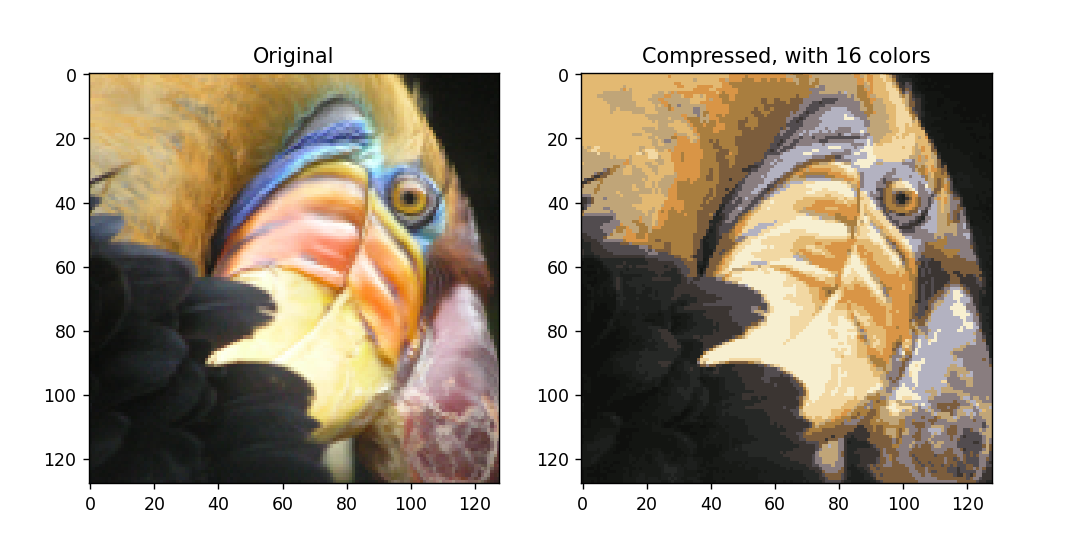
print('在图像的像素上运行K均值聚类。')
image = io.imread('bird_small.png')
image = img_as_float(image)
img_shape = image.shape
X = image.reshape(img_shape[0] * img_shape[1], 3)
K = 16
max_iters = 10
initial_centroids = kmic.kmeans_init_centroids(X, K)
centroids, idx = km.run_kmeans(X, initial_centroids, max_iters, False)
print('K均值完成。')
input('程序暂停。按回车键继续')
|
这里需要完成kMeansInitCentroids.py随机初始化质心的代码
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
| import numpy as np
def kmeans_init_centroids(X, K):
centroids = np.zeros((K, X.shape[1]))
random_indices = np.random.choice(X.shape[0], K, replace=False)
centroids = X[random_indices]
return centroids
|
random_indices = np.random.choice(X.shape[0], K, replace=False)的意思是:从 0 到 X.shape[0] - 1 的范围中,随机选择 K 个不重复的整数,用于初始化 K-Means 算法的质心。这样确保了初始化的质心是不同的样本点。
压缩图像
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
|
print('将 K-Means 应用于图像压缩。')
idx = fc.find_closest_centroids(X, centroids)
print(idx)
idx = np.array(idx, dtype=int)
print(idx)
X_recovered = centroids[idx]
X_recovered = np.reshape(X_recovered, (img_shape[0], img_shape[1], 3))
plt.subplot(2, 1, 1)
plt.imshow(image)
plt.title('Original')
plt.subplot(2, 1, 2)
plt.imshow(X_recovered)
plt.title('Compressed, with {} colors'.format(K))
plt.show()
input('练习 7 完成。按 ENTER 键退出')
|
输出图像
后半
概述
在这个练习中,你将使用主成分分析(PCA)进行降维操作。你将首先尝试使用一个2D示例数据集,以便直观地理解PCA的工作原理,然后将其应用于一个包含5000张人脸图像的较大数据集。
必要的文件:
ex7_pca.m - 用于主成分分析(PCA)第二练习的Python脚本
ex7data1.mat - PCA的示例数据集
ex7faces.mat - 人脸数据集
feature_normalize.py-特征归一化
需要补充的文件
pca.py - 执行主成分分析
projectData.py- 将数据集投影到较低维度的空间
recoverData.py - 从投影中恢复原始数据
导入
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
|
import matplotlib.pyplot as plt
import numpy as np
import scipy.io as scio
from mpl_toolkits.mplot3d import Axes3D
from skimage import io
from skimage.util import img_as_float
import featureNormalize as fn
import pca as pca
import runkMeans as rk
import projectData as pd
import recoverData as rd
import displayData as disp
import kMeansInitCentroids as kmic
import runkMeans as km
|
载入并绘图
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
print('可视化主成分分析示例数据集.')
data = scio.loadmat('ex7data1.mat')
X = data['X']
plt.figure()
plt.scatter(X[:, 0], X[:, 1], facecolors='none', edgecolors='b', s=20)
plt.axis('equal')
plt.axis([0.5, 6.5, 2, 8])
input('程序暂停。按回车键继续')
|
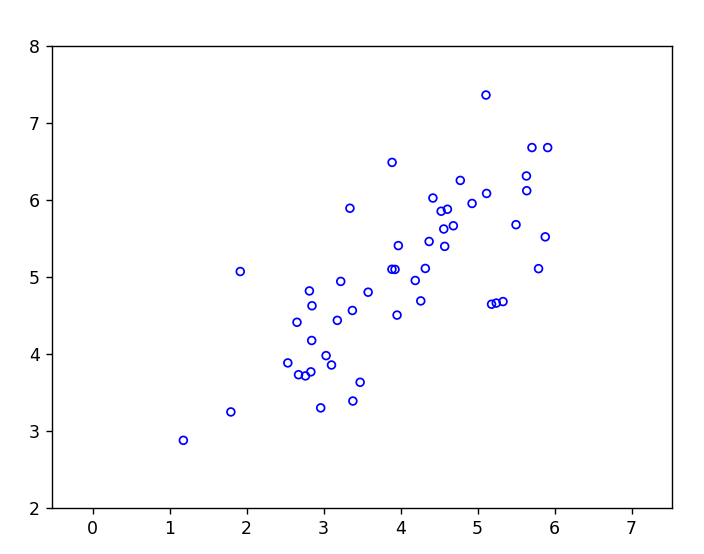
主成分分析
编写PCA的函数,根据PCA的公式:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
| import numpy as np
import scipy
def pca(X):
(m, n) = X.shape
U = np.zeros(n)
S = np.zeros(n)
Sigma = (1 / m) * X.T @ X
U, S, _ = scipy.linalg.svd(Sigma)
return U, S
|
回到ex7_pca.py文件
1
2
3
4
5
6
7
8
9
10
11
12
13
14
|
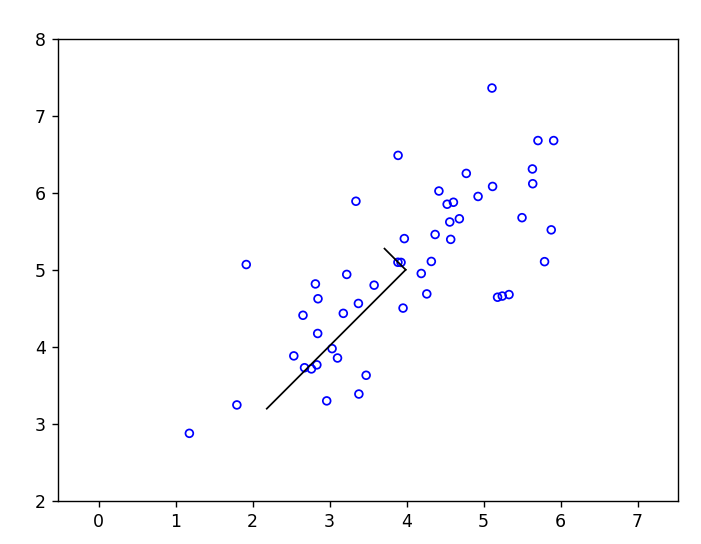
X_norm, mu, sigma = fn.feature_normalize(X)
U, S = pca.pca(X_norm)
rk.draw_line(mu, mu + 1.5 * S[0] * U[:, 0])
rk.draw_line(mu, mu + 1.5 * S[1] * U[:, 1])
plt.show()
print('最大特征向量: \nU[:, 0] = {}'.format(U[:, 0]))
print('预期结果应为 [-0.707107 -0.707107]')
input('程序暂停。按回车键继续')
|
输出图片:
数据降维
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
|
print('在示例数据集上进行降维.')
plt.figure()
plt.scatter(X_norm[:, 0], X_norm[:, 1], facecolors='none', edgecolors='b', s=20)
plt.axis('equal')
plt.axis([-4, 3, -4, 3])
K = 1
Z = pd.project_data(X_norm, U, K)
print('第一个示例的投影: {}'.format(Z[0]))
print('(此值应约为1.481274)')
|
先归一化,然后编写project_data函数,对数据进行降维
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
| import numpy as np
def project_data(X, U, K):
Z = np.zeros((X.shape[0], K))
for i in range(X.shape[0]):
x = X[i, :].reshape(X.shape[1], 1)
Z[i, :] = (U[:, :K].T@x).flatten()
return Z
|
x = X[i, :].reshape(X.shape[1], 1):这行代码将数据矩阵 X 中的第 i 行提取出来,并将其转换为列向量
Z[i, :] = (x.T @ U[:, :K]).flatten():这行代码执行以下操作:
(U[:, :K].T@x):将 x 与前 K 个主成分进行内积运算,得到投影后的列向量。这个操作实际上就是将数据投影到前 K 个主成分上。.flatten():将投影后的列向量展平成一维数组。Z[i, :] = ...:将得到的一维数组存储在矩阵 Z 的第 i 行中,表示第 i 个样本在前 K 个主成分上的投影。
数据复原
编写recover_data.py,将降维后的数据还原
$z=U^{T}_{reduce}x$,相反的方程为:$x_{appox}=U_{reduce}\cdot z$,$x_{appox}\approx x$
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
| import numpy as np
def recover_data(Z, U, K):
X_rec = np.zeros((Z.shape[0], U.shape[0]))
for i in range(Z.shape[0]):
v = Z[i, :].reshape(Z.shape[1], 1)
for j in range(U.shape[0]):
X_rec[i, j] = (v.T @ U[j, :K].T).flatten()
return X_rec
|
回到ex7_pca.py文件
1
2
3
4
5
6
7
8
9
10
11
| er_data(Z, U, K)
print('第一个示例的近似值: {}'.format(X_rec[0]))
print('(此值应约为 [-1.047419 -1.047419])')
plt.scatter(X_rec[:, 0], X_rec[:, 1], facecolors='none', edgecolors='r', s=20)
for i in range(X_norm.shape[0]):
rk.draw_line(X_norm[i], X_rec[i])
plt.figure()
plt.show()
|
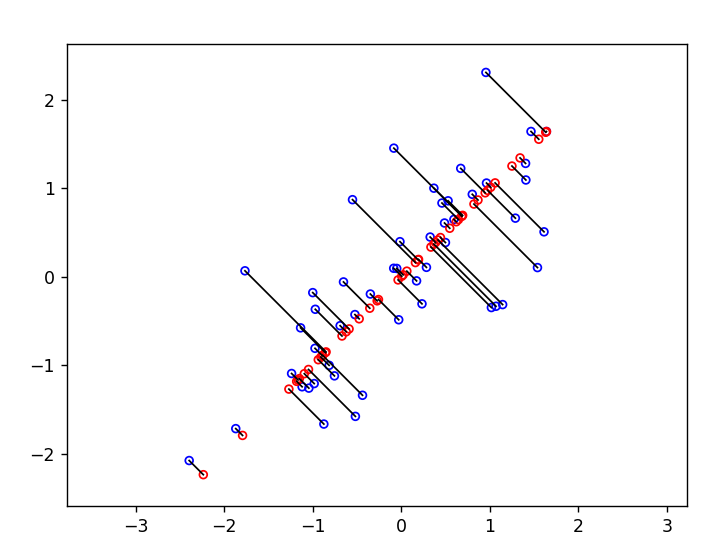
导入面部数据
1
2
3
4
5
6
7
8
9
10
11
12
13
|
print('加载面部数据集.')
data = scio.loadmat('ex7faces.mat')
X = data['X']
disp.display_data(X[0:100])
input('程序暂停。按回车键继续')
|

面部数据PCA
1
2
3
4
5
6
7
8
9
10
11
12
13
14
|
print('在面部数据集上运行PCA。\n(可能需要一两分钟...)')
X_norm, mu, sigma = fn.feature_normalize(X)
U, S = pca.pca(X_norm)
disp.display_data(U[:, 0:36].T)
input('程序暂停。按回车键继续')
|

面部数据降维
1
2
3
4
5
6
7
8
9
10
11
|
print('对面部数据进行降维.')
K = 100
Z = pd.project_data(X_norm, U, K)
print('投影数据Z的形状为: {}'.format(Z.shape))
input('程序暂停。按回车键继续')
|
可视化:
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
|
print('可视化投影后(降维后)的面部图像.')
K = 100
X_rec = rd.recover_data(Z, U, K)
disp.display_data(X_norm[0:100])
plt.title('原始面部图像',fontproperties='SimHei')
plt.axis('equal')
disp.display_data(X_rec[0:100])
plt.title('恢复后的面部图像',fontproperties='SimHei')
plt.axis('equal')
plt.show()
input('程序暂停。按回车键继续')
|

PCA可视化
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
|
image = io.imread('bird_small.png')
image = img_as_float(image)
img_shape = image.shape
X = image.reshape((img_shape[0] * img_shape[1], 3))
K = 16
max_iters = 10
initial_centroids = kmic.kmeans_init_centroids(X, K)
centroids, idx = km.run_kmeans(X, initial_centroids, max_iters, False)
selected = np.random.randint(X.shape[0], size=1000)
cm = plt.cm.get_cmap('RdYlBu')
fig = plt.figure()
ax = fig.add_subplot(111, projection='3d')
ax.scatter(X[selected, 0], X[selected, 1], X[selected, 2], c=idx[selected].astype(np.float64), s=15, cmap=cm, vmin=0, vmax=K)
plt.title('3D中绘制的像素数据集。颜色表示质心成员关系',fontproperties='SimHei')
plt.figure()
plt.show()
input('程序暂停。按回车键继续')
|
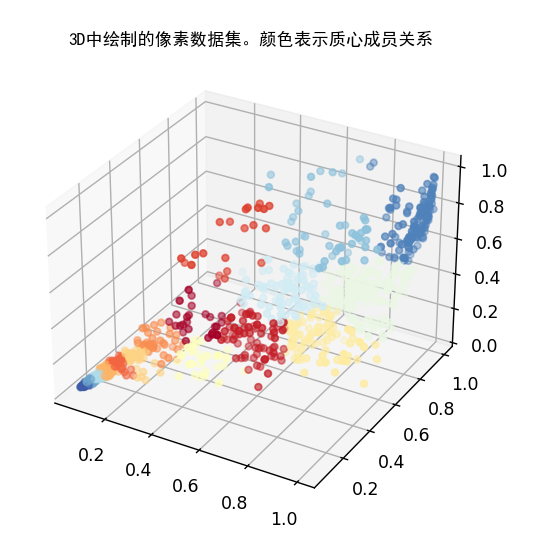
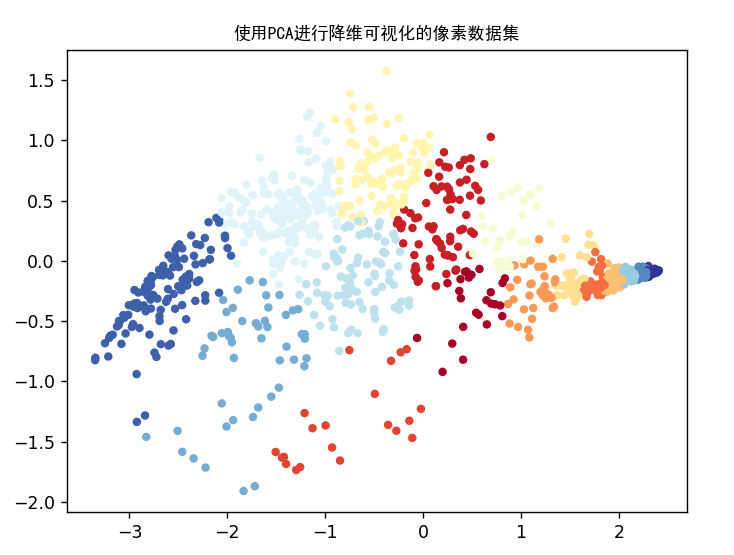
转为2D
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
|
X_norm, mu, sigma = fn.feature_normalize(X)
U, S = pca.pca(X_norm)
Z = pd.project_data(X_norm, U, 2)
plt.figure()
plt.scatter(Z[selected, 0], Z[selected, 1], c=idx[selected].astype(np.float64), s=15, cmap=cm)
plt.title('使用PCA进行降维可视化的像素数据集',fontproperties='SimHei')
plt.figure()
plt.show()
input('ex7_pca 完成。按回车键退出')
|